1 试验材料
试验在上海市农业科学院青浦白鹤试验基地石蒜种质资源圃进行。供试材料石蒜[Lycoris radiata(L’Her.)Herb]于 2016 年引进自南京中山植物园,为多年生开花种球。
由于引进的材料为野生种,不适合直接用于研究。因此于 2016—2018 年间培育了一批无性系材料。首先挑选 1 个多年生未分球且无病害的鳞茎作为母球,以鳞茎基盘为中心,按照“+”形平均切成 4 等分,进行扦插繁殖,获得了一批来自同一个母球的小鳞茎,并栽植于基质槽中,随后在不同的时间点选取部分小鳞茎继续进行鳞茎块扦插繁殖,最终获得一批同一遗传背景下不同年龄的石蒜小鳞茎,做好记录后栽植于同一基质槽中,进行一致的田间管理。
由于石蒜鳞茎的自然膨大过程较为缓慢,无法在一个生育周期内观察到鳞茎明显的发育过程,所以选择同一栽培环境条件下不同年龄的石蒜种球作为研究鳞茎膨大过程的材料。
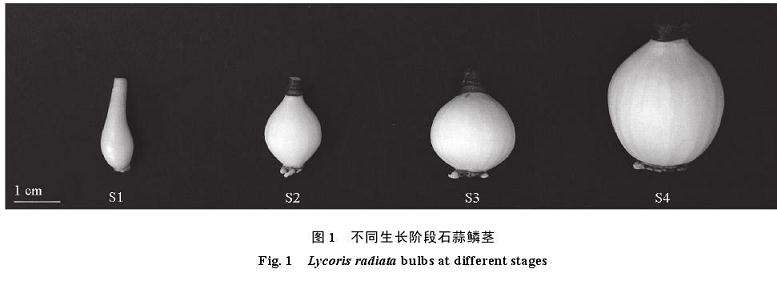
石蒜的生长周期较为独特,在气温升高时叶片枯萎(约 5 月初),并经历长达 5 个月的休眠期,直至花葶(8 月底—9 月初)和叶片(10 月底)相继抽出。本试验所选用的材料均为非开花球。2019年 1 月,石蒜进入营养生长旺期,此时叶片已完全抽出,挖取不同生长阶段的未分球的鳞茎,每个生长阶段取 6 个大小一致的种球(2 个为 1 次重复,设 3 次重复),去叶、去根、去除鳞茎褐色皮膜,清洗干净晾干后,记录鳞茎直径、周径和鲜样质量(图 1 和表 1),液氮速冻,–80 ℃冻存,备用于内源激素含量测定和转录组测序。
1.2 内源激素含量测定
利用外标法对内源激素含量进行定量,玉米素(zeatin,Z)、玉米素核苷(zeatin riboside,ZR)、3–吲哚乙酸(indole 3 acetic acid,IAA)、GA、ABA 标样均由美国 Sigma 公司生产,纯度 ≥ 98%。配置标准溶液时,分别称取 Z、ZR、IAA、GA 和 ABA,精确至 0.1 mg,用甲醇︰水(体积比为 1︰1)配制成 1 mg · m L-1的储备液,超声混匀。将 5 种标准储备液等体积混合配制成质量浓度 200 μg · m L-1的混合标准储备溶液,并逐级稀释为标准工作溶液,待上机检测。
石蒜鳞茎中内源激素的提取按照 Dobrev 和 Kaminek(2002)的方法进行。将样品在液氮中研磨成粉末后按 5 m L · g-1 FW 的比例加入–20 ℃预冷的提取液(甲醇︰水︰甲酸 = 15︰4︰1,体积比),–20 ℃放置 6 h 后 20 000 ? g 离心 15 min。收集上清液后,重复以上操作,–20 ℃放置 30 min 后离心。合并上清液,过预先用 5 m L 甲醇和 5 m L 1 mol · L-1甲酸处理过的 C-18 固相萃取柱。将过柱后的样品转入 25 m L 烧杯中,冷冻真空浓缩干燥。
干燥后用 5 m L 甲醇复溶样品,过 0.45 μm 微孔滤膜后用高效液相色谱法(HPLC)分析内源激素含量(高效液相色谱仪型号为 Agilent 1100):流动相成分为体积浓度 0.1%的乙酸与甲醇体积比为6︰4。进样量 10 μL,流速 0.8 m L · min-1,柱温 30 ℃,走样时间为 40 min,检测波长 254 nm。在选定的色谱条件下对混合的标准样品进行 HPLC 检测。数据用 Agilent 公司软件进行分析。
1.3 石蒜鳞茎 RNA 的提取及反转录
使用 Ta Ka Ra 公司的 Mini BEST Plant RNA Extraction Kit(Code No. 9769)提取石蒜鳞茎中总RNA,并用 DNase I(Ambion,USA)处理去除基因组 DNA,使用 Nano Drop 2000 超微量分光光度计(Thermo Fisher Scientific,USA)和 1%凝胶电泳分别检测 RNA 质量和完整性,OD260/280 = 1.8 ~ 2.2的 RNA 被用于转录组测序建库及后续表达分析研究。


1.4 c DNA 文库构建与测序
c DNA 文库的构建参考谭嫣等(2019)的方法进行。首先用带有 Olig(d T)的磁珠富集 m RNA,随机打断后,利用六碱基(N6)随机引物反转录成 c DNA 第一链,并以其为模板合成双链 c DNA,随后对其进行粘性末端修复加 A 尾并连接测序接头,用 AMPure XP beads 进行片段大小选择,最后进行 PCR 富集得到最终的 c DNA 文库,利用深圳华大基因科技发展有限公司的 BGISEQ-500 平台进行测序分析。

1.5 转录组数据分析
首先对原始 Raw reads 数据进行过滤,去除读取质量低的序列,得到高质量的 Clean reads,利用默认参数下的 Trinity 软件将 Clean reads 组装成 Unigene(Xi et al.,2019)。Unigene 的表达水平基于 FPKM(Fragments Per Kilobase Million)值来进行计算。使用 NOISeq 方法(Tarazona et al.,2011)筛选生物学重复样品间的差异表达基因,其筛选条件则为 FDR(false discovery rate)< 0.05 以及|log2 fold change| ≥ 1。
Unigene 序列在核苷酸和蛋白数据库中比对同源性,包括 Nr 数据库、Swiss Prot 蛋白数据库、KEGG 代谢通路数据库、KOG/COG 数据库等。基于比对结果使用 Gene ontology 数据库对所鉴定的基因进行功能注释。

1.6 实时荧光定量 PCR(q RT-PCR)
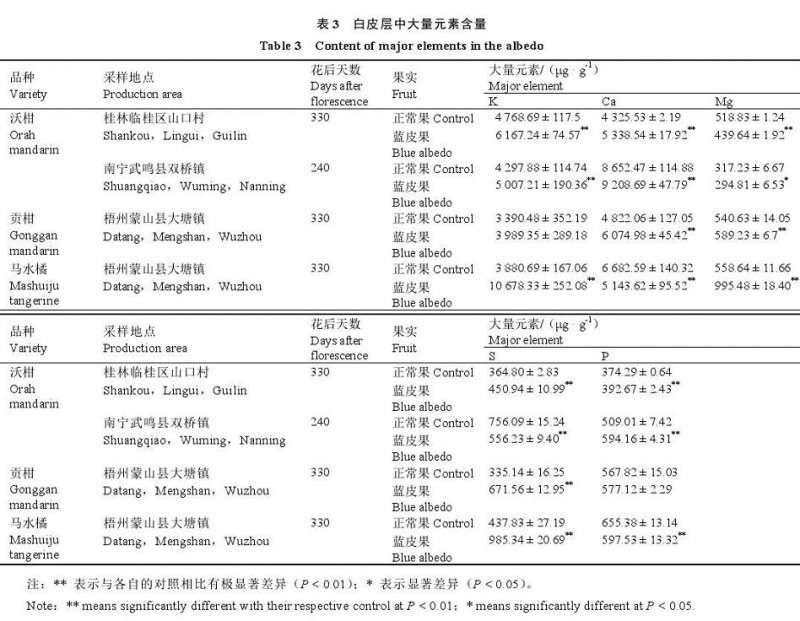
按照 Prime scriptTM RT Reagent Kit with g DNAEraser 试剂盒(Ta Ka Ra,中国)说明书将 RNA 进行反转录,合成 c DNA。按照 TB GreenTM Premix Ex TaqTMⅡ(Ta Ka Ra,中国)试剂盒和 ABI 7500荧光定量 PCR 仪进行 q RT-PCR 反应,以 Actin 作为内标基因,各基因引物序列如表 2 所示。

